ブレーカが飛び,BuffaloのLS-QL/1Dが壊れた.
ディスクは生きていたけど,結構ショック.
なかなか気に入ってたのにサ...
泣いていても仕方がないので後継機を買いにビックカメラへ.
色々と悩んだあげく,
裸族のインテリジェントビルNAS(CRIB35NAS)を購入した.
作りがしっかりしてたのと,NFSにも対応していたのが決めてになった.
本当は自分で組むべきなのかもしれないけど,
色々と面倒だし,パッケージングされている方が家族も使いやすい.
buffaloのディスクからmount -t xfsでデータを取り出し,
運用開始.
さあ,どうなることやら.
とりあえず,長生きしてね.
2011年12月19日月曜日
2011年12月17日土曜日
Apache POI:Not enough data (1) to read requested (2) bytes
Apache POIライブラリ(3.7)でxlsファイルを読み込んだら次のようなエラーが表示された.
気持ち悪いが,とりあえず動いたからよしとしよう.
Exception in thread "main" org.apache.poi.hssf.record.RecordFormatException: Unable to construct record instance原因がわからずなんともならなかったのでベータ版(3.8beta4)に切り替えたら,あっさりと動いた.
at org.apache.poi.hssf.record.RecordFactory$ReflectionConstructorRecordCreator.create(RecordFactory.java:65)
at org.apache.poi.hssf.record.RecordFactory.createSingleRecord(RecordFactory.java:300)
at org.apache.poi.hssf.record.RecordFactoryInputStream.readNextRecord(RecordFactoryInputStream.java:270)
at org.apache.poi.hssf.record.RecordFactoryInputStream.nextRecord(RecordFactoryInputStream.java:236)
at org.apache.poi.hssf.record.RecordFactory.createRecords(RecordFactory.java:442)
at org.apache.poi.hssf.usermodel.HSSFWorkbook.<init>(HSSFWorkbook.java:263)
at org.apache.poi.hssf.usermodel.HSSFWorkbook.<init>(HSSFWorkbook.java:188)
at org.apache.poi.hssf.usermodel.HSSFWorkbook.<init>(HSSFWorkbook.java:305)
at org.apache.poi.hssf.usermodel.HSSFWorkbook.<init>(HSSFWorkbook.java:286)
at org.apache.poi.ss.usermodel.WorkbookFactory.create(WorkbookFactory.java:60)
at org.sheepcloud.lab.Parser.<init>(Parser.java:26)
at org.sheepcloud.lab.Parser.main(Parser.java:53)
Caused by: org.apache.poi.hssf.record.RecordFormatException: Not enough data (1) to read requested (2) bytes
at org.apache.poi.hssf.record.RecordInputStream.checkRecordPosition(RecordInputStream.java:216)
at org.apache.poi.hssf.record.RecordInputStream.readUShort(RecordInputStream.java:267)
at org.apache.poi.hssf.record.common.UnicodeString$PhRun.<init>(UnicodeString.java:311)
at org.apache.poi.hssf.record.common.UnicodeString$PhRun.<init>(UnicodeString.java:297)
at org.apache.poi.hssf.record.common.UnicodeString$ExtRst.<init>(UnicodeString.java:178)
at org.apache.poi.hssf.record.common.UnicodeString.<init>(UnicodeString.java:438)
at org.apache.poi.hssf.record.SSTDeserializer.manufactureStrings(SSTDeserializer.java:55)
at org.apache.poi.hssf.record.SSTRecord.<init>(SSTRecord.java:250)
at sun.reflect.NativeConstructorAccessorImpl.newInstance0(Native Method)
at sun.reflect.NativeConstructorAccessorImpl.newInstance(NativeConstructorAccessorImpl.java:39)
at sun.reflect.DelegatingConstructorAccessorImpl.newInstance(DelegatingConstructorAccessorImpl.java:27)
at java.lang.reflect.Constructor.newInstance(Constructor.java:513)
at org.apache.poi.hssf.record.RecordFactory$ReflectionConstructorRecordCreator.create(RecordFactory.java:57)
... 11 more
気持ち悪いが,とりあえず動いたからよしとしよう.
2011年12月4日日曜日
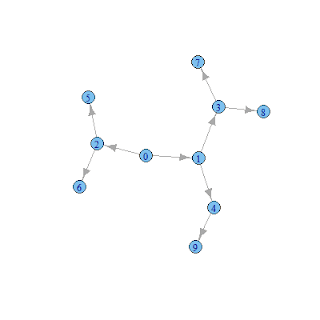
ダイクストラ法
ダイクストラ法をRとigraphを使って表現する.
igraphのshortest.paths関数で一発でできるのだけど,お勉強のためということで.
まずは最短経路を求めるグラフを用意する.
library(igraph)
g<-graph.empty(5)
V(g)$name<-c("A","B","C","D","E")
g<-add.edges(g,c(0,1,0,2,0,3,1,2,1,3,1,4,2,3,3,4))
g<-as.undirected(g)
E(g)$weight<-c(2,1,7,5,6,1,1,8)
lay<-rbind(c(1,3),c(2,3),c(1,1),c(2,1),c(3,2))
plot(g, layout=lay,vertex.label=V(g)$name,vertex.size=12,edge.label=E(g)$weight)
こんな感じ.今回はノード「E」をゴールとする.
つぎに各ノードのコストを初期化する.
V(g)$cost<-c(Inf,Inf,Inf,Inf,0)
V(g)$fixed=FALSE
V(g)$color<-"skyblue"
plot(g, layout=lay,vertex.label=V(g)$cost,vertex.size=12,edge.label=E(g)$weight,vertex.color=V(g)$color)
つぎはコスト計算.
V(g)[nei(V(g)[fixed==FALSE & cost==min(V(g)[fixed==FALSE]$cost)]) & fixed==FALSE][V(g)[nei(V(g)[fixed==FALSE & cost==min(V(g)[fixed==FALSE]$cost)]) & fixed==FALSE]$cost>(E(g)[V(g)[fixed==FALSE] %->% V(g)[fixed==FALSE & cost==min(V(g)[fixed==FALSE]$cost)]]$weight)+(V(g)[fixed==FALSE & cost==min(V(g)[fixed==FALSE]$cost)]$cost)]$cost<-(E(g)[V(g)[fixed==FALSE] %->% V(g)[fixed==FALSE & cost==min(V(g)[fixed==FALSE]$cost)]]$weight)+(V(g)[fixed==FALSE & cost==min(V(g)[fixed==FALSE]$cost)]$cost)ながい...もっと短くできるかもしれない.
V(g)[fixed==FALSE & cost==min(V(g)[fixed==FALSE]$cost)]$color="yellow"計算が終わったノードは黄色に塗りつぶす.
V(g)[fixed==FALSE & cost==min(V(g)[fixed==FALSE]$cost)]$fixed=TRUE
plot(g, layout=lay,vertex.label=V(g)$cost,vertex.size=12,edge.label=E(g)$weight,vertex.color=V(g)$color)
これを計算するノードが無くなるまで繰り返す.
もう一回.
もう一回.
もう一回.
もう一回.
これで終わり.
実際にグラフを描きながらだと楽しいね.
2011年12月1日木曜日
R + igraph + Cytoscape
まずはR+igraphでネットワークグラフを作成する.
> library(igraph)
> d<-matrix(c(0,0,1,1,1 ,0,1,1,0,2,0,3,1,0,1,6,0,0,0,2,1,8,5,2,0),ncol=5,nrow=5)
> ls()
[1] "d"
> d
[,1] [,2] [,3] [,4] [,5]
[1,] 0 0 0 6 1
[2,] 0 1 3 0 8
[3,] 1 1 1 0 5
[4,] 1 0 0 0 2
[5,] 1 2 1 2 0
> g<-graph.adjacency(d,weighted=TRUE)
> summary(g)
Vertices: 5
Edges: 15
Directed: TRUE
No graph attributes.
No vertex attributes.
Edge attributes: weight.
> degree(g)
[1] 5 6 7 4 8
> png("c:/tmp/myplot.png",width=600, height=600,pointsize=14)
> plot(g,layout=layout.fruchterman.reingold,vertex.label=V(g)$smr,vertex.size=degree(g)*4)
> dev.off()
null device
1
なかなか,いい感じ.
つぎはCytoscape(http://www.cytoscape.org/)を使って描画してみる.
まずは,Rからグラフをエキスポートする.
このときファイルタイプとして”ncol”を指定するのを忘れずに!
> write.graph(g,"c:/tmp/mygraph.ncol","ncol")
Cytoscapeを起動して[File]>[Import]>[Network from Table(Text/MS Excel...)]を選択する.
先ほどエキスポートした「c:/tmp/mygraph.ncol」を読み込む.
[Source Interaction]に「Column 1」,[Interaction Type]に「Column 3」,[Target Interaction]に「Column 2」を設定する.
[Import]ボタンをクリックする.
これでグラフをインポートできた.
あとはCytoscpae上でグラフを色々と見やすくしてやればいい.
2011年11月27日日曜日
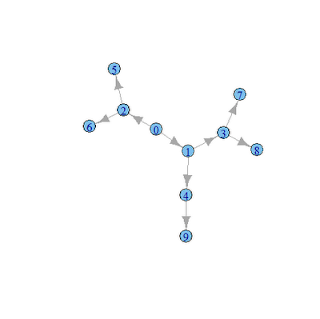
R+igraphのお勉強 その3
ノードの次数を確認する.
入次数を確認する.
> g次数を確認する.
Vertices: 10
Edges: 9
Directed: TRUE
Edges:
[0] 0 -> 1
[1] 0 -> 2
[2] 1 -> 3
[3] 1 -> 4
[4] 2 -> 5
[5] 2 -> 6
[6] 3 -> 7
[7] 3 -> 8
[8] 4 -> 9
> degree(g)
[1] 2 3 3 3 2 1 1 1 1 1
入次数を確認する.
> degree(g,mode="in")出次数を確認する.
[1] 0 1 1 1 1 1 1 1 1 1
> degree(g,mode="out")当然,無向グラフに入出の区別はない.
[1] 2 2 2 2 1 0 0 0 0 0
> g2次数を確認する.
Vertices: 10
Edges: 9
Directed: FALSE
Edges:
[0] 0 -- 1
[1] 0 -- 2
[2] 1 -- 3
[3] 1 -- 4
[4] 2 -- 5
[5] 2 -- 6
[6] 3 -- 7
[7] 3 -- 8
[8] 4 -- 9
> degree(g2)ノードの次数をノードのサイズに設定してグラフを描画する.
[1] 2 3 3 3 2 1 1 1 1 1
> degree(g2)
[1] 2 3 3 3 2 1 1 1 1 1
> degree(g2,mode="in")
[1] 2 3 3 3 2 1 1 1 1 1
> degree(g2,mode="out")
[1] 2 3 3 3 2 1 1 1 1 1
> png("plot3.png",width=400, height=400,pointsize=12)次数の10倍をノードサイズとする.
> plot(g,layout=lay,vertex.size=degree(g)*10)
> dev.off()
null device
1
R+igraphのお勉強 その2
前回の続き.
有向グラフを無向グラフに変換してみる.
有向グラフの確認.
作業経過をファイル保存する.
有向グラフを無向グラフに変換してみる.
有向グラフの確認.
> is.directed(g)無向グラフに変換する.
[1] TRUE
> g2<-as.undirected(g)変換結果を確認する.
> g2グラフを描画する.
Vertices: 10
Edges: 9
Directed: FALSE
Edges:
[0] 0 -- 1
[1] 0 -- 2
[2] 1 -- 3
[3] 1 -- 4
[4] 2 -- 5
[5] 2 -- 6
[6] 3 -- 7
[7] 3 -- 8
[8] 4 -- 9
> is.directed(g2)
[1] FALSE
> png("plot2.png",width=400, height=400,pointsize=12)
> plot(g2,layout=lay)
> dev.off()
null device
1
> save(g,g2,lay,file="test.dat")
R+igraphのお勉強 その1
igraphライブラリを読み込む.
> library(igraph)10ノードのツリーグラフを作成する.
> g<-graph.tree(10)グラフをレイアウトする.
> summary(g)
Vertices: 10
Edges: 9
Directed: TRUE
No graph attributes.
No vertex attributes.
No edge attributes.
> g
Vertices: 10
Edges: 9
Directed: TRUE
Edges:
[0] 0 -> 1
[1] 0 -> 2
[2] 1 -> 3
[3] 1 -> 4
[4] 2 -> 5
[5] 2 -> 6
[6] 3 -> 7
[7] 3 -> 8
[8] 4 -> 9
> lay<-layout.fruchterman.reingold(g)
> layここまでの作業をファイル保存する.
[,1] [,2]
[1,] 0.5750165 -0.7339218
[2,] 4.2972271 -0.9157740
[3,] -2.8644359 0.1233019
[4,] 5.7313203 2.7760283
[5,] 5.3732637 -4.4586431
[6,] -3.4715509 3.4824008
[7,] -4.1404984 -2.9629157
[8,] 4.2804038 5.9832150
[9,] 8.9190178 2.4068283
[10,] 4.1109849 -7.2698722
> save(g,lay,file="test.dat")グラフを描画する.
> plot(g,layout=lay)
> png("plot.png",width=400, height=400,pointsize=12)
> plot(g,layout=lay)
> dev.off()
null device
1
2011年11月20日日曜日
グラフレイアウト
とりあえずライブラリをロードする.
グラフをレイアウトする.
> library(igraph)とりあえずツリーグラフを作成する.
> g<-graph.tree(4)
> plot.igraph(g)
> lay<-layout.kamada.kawai(g)再描画.
> lay
[,1] [,2]
[1,] -1.95786789 -0.6726298
[2,] -0.97281249 -1.2272012
[3,] -2.94060962 -0.1762201
[4,] 0.09102538 -0.7586657
[5,] -1.03996471 -2.3633110
[6,] -3.22145269 0.8896139
[7,] -3.96005388 -0.5896342
[8,] 0.59269741 0.2250630
[9,] 1.10354845 -1.2044752
[10,] -1.05079882 -3.4245322
> plot.igraph(g,layout=lay)
ちゃんとレイアウトされた.
2011年11月19日土曜日
Rでネットワーク図を描く3
今度は行ラベル,列ラベルを付けてみる.
エッジの重みを表示する.
今度は特定のエッジを削除してみる.
重みが5以下のエッジを削除してみよう.
次数0のノードができてしまった.
こいつを削除しよう.
ちゃんと削除できた.
> d<-matrix(c(0,0,1,1,1 ,0,1,1,0,2,0,3,1,0,1,6,0,0,0,2,1,8,5,2,0),ncol=5,nrow=5まずは列ラベルを設定する.
> d
[,1] [,2] [,3] [,4] [,5]
[1,] 0 0 0 6 1
[2,] 0 1 3 0 8
[3,] 1 1 1 0 5
[4,] 1 0 0 0 2
[5,] 1 2 1 2 0
> colnames(d)<-c("A","B","C","D","E")お次は行ラベルを設定する.
> d
A B C D E
[1,] 0 0 0 6 1
[2,] 0 1 3 0 8
[3,] 1 1 1 0 5
[4,] 1 0 0 0 2
[5,] 1 2 1 2 0
> rownames(d)<-c("A","B","C","D","E")グラフに変換する.
> d
A B C D E
A 0 0 0 6 1
B 0 1 3 0 8
C 1 1 1 0 5
D 1 0 0 0 2
E 1 2 1 2 0
> g<-graph.adjacency(d,weighted=TRUE)画像に書き出す.
> png("c:/tmp/myplot.png",width=400, height=400,pointsize=12)
> plot(g)
> dev.off()
null device
1
エッジの重みを表示する.
> E(g)$weight一応,グラフ情報もみておく.
[1] 6 1 1 3 8 1 1 1 5 1 2 1 2 1 2
> summary(g)
Vertices: 5
Edges: 15
Directed: TRUE
No graph attributes.
Vertex attributes: name.
Edge attributes: weight.
今度は特定のエッジを削除してみる.
重みが5以下のエッジを削除してみよう.
> g2<-delete.edges(g,E(g)[weight<=5])グラフ情報をみると,
> summary(g2)描画してみよう.
Vertices: 5
Edges: 2
Directed: TRUE
No graph attributes.
Vertex attributes: name.
Edge attributes: weight.
> png("c:/tmp/myplot.png",width=400, height=400,pointsize=12)
> plot(g2,layout=layout.fruchterman.reingold,vertex.color="white",vertex.label=V(g2)$name,edge.label=E(g2)$weight,vertex.size=10)
> dev.off()
null device
1
次数0のノードができてしまった.
こいつを削除しよう.
> g2<-delete.vertices(g2,which(degree(g2)<1)-1)もう一回,書き出してみる.
> summary(g2)
Vertices: 4
Edges: 2
Directed: TRUE
No graph attributes.
Vertex attributes: name.
Edge attributes: weight.
> png("c:/tmp/myplot.png",width=400, height=400,pointsize=12)
> plot(g2,layout=layout.fruchterman.reingold,vertex.color="white",vertex.label=V(g2)$name,edge.label=E(g2)$weight,vertex.size=10)
> dev.off()
null device
1
ちゃんと削除できた.
2011年11月18日金曜日
Rでネットワーク図を描く2
こんどはもう少し大きめのグラフを書いてみる.
重みをエッジの幅に設定する.
> library(igraph)
> d<-matrix(c(0,0,1,1,1 ,0,1,1,0,2,0,3,1,0,1,6,0,0,0,2,1,8,5,2,0),ncol=5,nrow=5
> d
[,1] [,2] [,3] [,4] [,5]
[1,] 0 0 0 6 1
[2,] 0 1 3 0 8
[3,] 1 1 1 0 5
[4,] 1 0 0 0 2
[5,] 1 2 1 2 0
> g<-graph.adjacency(d,weighted=TRUE)
> png("c:/tmp/myplot.png",width=400, height=400,pointsize=12)
> plot(g,,layout=layout.fruchterman.reingold,vertex.color="white",vertex.label=V(g)$name,edge.color="black",edge.label=E(g)$weight,vertex.size=10)
> dev.off()
null device
1
> E(g)$weight美しくないのでやっぱり戻そう.
[1] 6 1 1 3 8 1 1 1 5 1 2 1 2 1 2
> E(g)$width<-E(g)$weight
> E(g)$width<-1
Rでネットワーク図を描く
Rが面白い.簡単に色々とできてしまう.
今回はigraphライブラリでネットワーク図の作成にトライしてみる.
まずライブラリを読み込む.
今回はigraphライブラリでネットワーク図の作成にトライしてみる.
まずライブラリを読み込む.
> library(igraph)つぎに隣接行列を作る.
> d<-matrix(c(0,0,1,1,0,1,1,0,0),ncol=3,nrow=3)行列のサイズを確認する.
> dim(d)行列を表示する.
[1] 3 3
> d行列から重み付き有向グラフを作成する.
[,1] [,2] [,3]
[1,] 0 1 1
[2,] 0 0 0
[3,] 1 1 0
> g<-graph.adjacency(d,mode="directed",weighted=TRUE)グラフ情報を確認する.
> summary(g)最後にグラフを画像ファイルに書き出す.
Vertices: 3
Edges: 4
Directed: TRUE
No graph attributes.
No vertex attributes.
Edge attributes: weight.
> png("c:/tmp/myplot.png",width=400, height=400,pointsize=12)
> plot(g,vertex.color="white",vertex.label=V(g)$name,edge.color="black",edge.label=E(g)$weight,vertex.size=10)
> dev.off()
null device
1
2011年11月11日金曜日
Androidのスクリーンショットを撮る
1. USBデバッグモードをONにする.
2. ddmsを起動する.
4. メニュー「Device」>「Screen capture...」を選択する.
5. あとは画面に従って操作するだけ.
6. こんな風にスクリーンショット撮れる.便利だのう.
2. ddmsを起動する.
C:\android-sdk-windows\tools>ddms.bat3. スクリーンショットを撮りたいdeviceを選択する.
4. メニュー「Device」>「Screen capture...」を選択する.
5. あとは画面に従って操作するだけ.
6. こんな風にスクリーンショット撮れる.便利だのう.
Android SDKとGoogle USB Driverのインストールメモ
Androidを色々と弄りたくなってきた.
とりあえずSDKをインストールするところからはじめたいと思う.
以下は作業メモ.
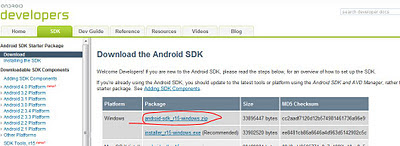
SDKのダウンロードサイト(http://developer.android.com/sdk/index.html)からzipファイルをダウンロードする.
ダウンロードしたファイルを適当な場所に展開する.
今回はC:\の直下に配置する.
あとは必要に応じてパスを通せばいい.
ついでにGoogle USB Driverもインストールしておこう.
SDK Manager.exeを起動する.
Extras>Google USB Driverにチェックをいれて「install package」をクリックする.
Google USB Driverはデフォルトで選択状態にならないのでAcceptを選択する.
あとはダウンロード&インストールが完了するまでしばらく待とう.
androidのUSBデバッグをONにしてPCと接続する.
デバイスマネージャを開き,「ほかのデバイス」に表示されている「ADB」のプロパティを開く.
「詳細」タブを選択し,プロパティの中からハードウェアIDを選択する.
ここの値「USB\VID_0BB4&PID_0C87&MI_01」(deviceにより異なる)をメモする.
「\android-sdk-windows\extras\google\usb_driver\android_winusb.inf」をメモ帳などで開く.
「[Google.NTx86]」に先ほどメモしたハードウェアIDを追加する.AMD系CPUなら「[Google.NTamd64]」に追加する.
あとはドライバを組み込んでandroidデバイスを再認識させてやるだけ.
Android PhoneとしてADB Interfaceが認識できればOK.
やった.
とりあえずSDKをインストールするところからはじめたいと思う.
以下は作業メモ.
SDKのダウンロードサイト(http://developer.android.com/sdk/index.html)からzipファイルをダウンロードする.
ダウンロードしたファイルを適当な場所に展開する.
今回はC:\の直下に配置する.
あとは必要に応じてパスを通せばいい.
ついでにGoogle USB Driverもインストールしておこう.
SDK Manager.exeを起動する.
Extras>Google USB Driverにチェックをいれて「install package」をクリックする.
Google USB Driverはデフォルトで選択状態にならないのでAcceptを選択する.
あとはダウンロード&インストールが完了するまでしばらく待とう.
androidのUSBデバッグをONにしてPCと接続する.
デバイスマネージャを開き,「ほかのデバイス」に表示されている「ADB」のプロパティを開く.
「詳細」タブを選択し,プロパティの中からハードウェアIDを選択する.
ここの値「USB\VID_0BB4&PID_0C87&MI_01」(deviceにより異なる)をメモする.
「\android-sdk-windows\extras\google\usb_driver\android_winusb.inf」をメモ帳などで開く.
「[Google.NTx86]」に先ほどメモしたハードウェアIDを追加する.AMD系CPUなら「[Google.NTamd64]」に追加する.
; HTC X06HT2 %SingleAdbInterface% = USB_Install, USB\VID_0BB4&PID_0C87 %CompositeAdbInterface% = USB_Install, USB\VID_0BB4&PID_0C87&MI_01以上でGoogle USB Driverの設定が完了した.
あとはドライバを組み込んでandroidデバイスを再認識させてやるだけ.
Android PhoneとしてADB Interfaceが認識できればOK.
やった.
GmailのPOP機能メモ
GmailのPOP機能では,メッセージをダウンロードできるのは1クライアントだけ.
POP機能を使って,複数クライアントでメッセージを共有することは基本できない.
https://mail.google.com/support/bin/answer.py?hl=ja&answer=13289&topic=1668962
ただし,Gmailの最新モードを使うとそれが可能になる.
最新モードだと,過去30日間のメッセージを複数のクライアントから,ダウンロードできるようになる.
最新モードで使うには,POPログインユーザ名を「username@gmail.com」を「recent:username@gmail.com」とするだけ.
https://mail.google.com/support/bin/answer.py?hl=ja&answer=47948&topic=1668962
IMAPの方がいいと思うけど,どうしてもPOPを使いたい人は知っておくと便利かも知れない.
POP機能を使って,複数クライアントでメッセージを共有することは基本できない.
https://mail.google.com/support/bin/answer.py?hl=ja&answer=13289&topic=1668962
ただし,Gmailの最新モードを使うとそれが可能になる.
最新モードだと,過去30日間のメッセージを複数のクライアントから,ダウンロードできるようになる.
最新モードで使うには,POPログインユーザ名を「username@gmail.com」を「recent:username@gmail.com」とするだけ.
https://mail.google.com/support/bin/answer.py?hl=ja&answer=47948&topic=1668962
IMAPの方がいいと思うけど,どうしてもPOPを使いたい人は知っておくと便利かも知れない.
Google Docs SpreadSheetグラフ機能のテスト
今更だけど,東京電力のでんき予報をSpreadSheetでグラフ化してみる.
データの取り込みはimportdataを使用する.
=importdata("http://www.tepco.co.jp/forecast/html/images/juyo-j.csv")
interactive chartで公開したかったけどエラーが発生.
しかたなくimageで公開してみる.
共有設定はAnyone with the linkで十分みたい.

更新タイミングが不明だけど,ちゃんと更新されている.
いい感じ.
データの取り込みはimportdataを使用する.
=importdata("http://www.tepco.co.jp/forecast/html/images/juyo-j.csv")
interactive chartで公開したかったけどエラーが発生.
しかたなくimageで公開してみる.
共有設定はAnyone with the linkで十分みたい.
更新タイミングが不明だけど,ちゃんと更新されている.
いい感じ.
登録:
投稿 (Atom)